Synthese ribosomaler RNA und Regulation der RNA-Polymerase-I-Transkription im Kontext von Chromatin in S. cerevisiae
Die Biogenese eukaryotischer Ribosomen beginnt mit der Synthese der ribosomalen (r)RNAs durch die RNA-Polymerasen (Pol) I und III. Während Pol III das 5S-rRNA-Gen transkribiert, transkribiert Pol I ein großes Vorläufermolekül, das die Sequenzen der übrigen drei rRNAs umfasst, in einem nukleären Subkompartiment, dem Nucleolus. Die Pol-I-Transkription und die Reifung der Ribosomen sind eng miteinander gekoppelt, da Faktoren der Ribosomenbiogenese und ribosomale Proteine bereits während der Transkription mit der entstehenden rRNA assembliert werden.
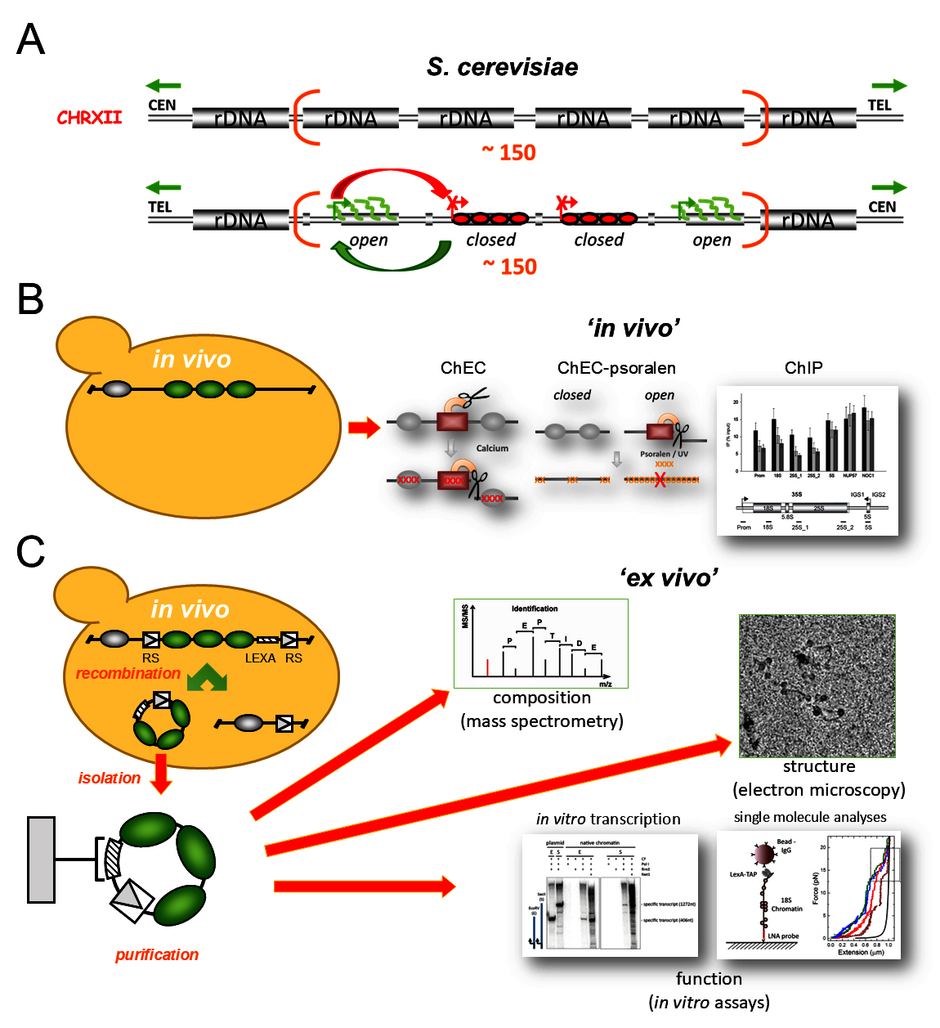
Obwohl Pol I nur einen genomischen Lokus transkribiert, ist das Enzym für über 60 % der zellulären RNA-Synthese in exponentiell wachsenden S. cerevisiae-Zellen (im Folgenden als Hefe bezeichnet) verantwortlich. Dies wird durch einen starken Promotor mit einem stabil gebundenen Präinitiationskomplex (PIC) sowie durch die Multimerisierung der rRNA-Gene an einer oder mehreren Stellen in eukaryotischen Genomen erreicht (Abbildung A). Selbst in sich teilenden Zellen werden nicht alle rRNA-Gene transkribiert, sondern existieren in mindestens zwei verschiedenen Chromatin-Zuständen nebeneinander. Während transkriptionsinaktive rRNA-Gene in Nukleosomen eingebaut sind und sich in einem sogenannten „geschlossenen“ Chromatin-Zustand befinden, liegen aktiv transkribierte rRNA-Gene in einem nukleosomenarmen „offenen“ Chromatin-Zustand vor. rRNA-Gene können je nach physiologischem Zustand der Zelle zwischen den beiden Chromatin-Zuständen wechseln.
Unsere Forschung zielt darauf ab, die molekularen Mechanismen zu identifizieren, die diesen Chromatin-Übergängen zugrunde liegen. Letztlich möchten wir zum Verständnis des Zusammenspiels zwischen der Chromatinstruktur der rRNA-Gene, der Pol-I-Transkription und der frühen Ribosomenreifung beitragen. Diesen komplexen Prozesse versuchen wir, zusammen mit der AG Engel und der AG Milkereit, besser zu verstehen. Zu diesem Zweck nutzen und entwickeln wir Techniken, die es uns ermöglichen, die Chromatinstruktur der rRNA-Gene und die Pol-I-Transkription sowohl in vivo (Abbildung B) als auch im nativen Chromatinkontext ex vitro zu untersuchen (Abbildung C).